Matematikere afslører smitteveje med corona
En gruppe kvantematematikere fra Københavns Universitet har sporet smitteveje af den nye coronavirus ved at analysere mutationer i danske coronavirussekvenser.
Af Matthias Christandl, Frederik Ravn Klausen, Vincent Steffan og Albert H. Werner
Da den nye coronavirus ramte hele verden, og i starten af marts også Danmark, var det en ekstraordinær begivenhed. Samfundet blev sat i alarmberedskab, og de fleste blev sendt hjem fra arbejde og skole. Situationen medførte også, at en masse data pludselig blev tilgængelig. Man kan stadig knap nok åbne en avis uden at blive konfronteret med grafer, der viser den seneste sygdomsudvikling. Det gjaldt også gendata om selve coronavirusset, hvor der allerede i marts fandtes flere sekvenser af virusgenomet end fra tidligere pandemier på grund af den rivende teknologiske udvikling inden for genforskningen.
Den store datamængde gjorde, at vi og vores kolleger ved det tværvidenskabelige forskningscenter QMATH ved Københavns Universitet Andreas Bluhm, Fulvio Gesmundo, Laura Mančinska, og Daniel Stilck França blev nysgerrige. Til daglig forsker vi i kvantematematik, men vi ville gerne forstå, hvad man kunne bruge det data til og se, om vores matematiske kundskaber kunne bidrage til at forstå pandemien.
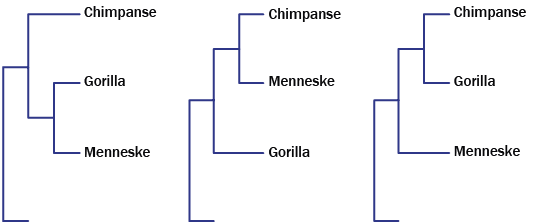
De genetiske data fortæller os blandt andet, at virussen med tiden får mutationer på mere eller mindre tilfældige steder. Er der opstået en mutation i virusgenomet hos én inficeret person, bliver den videregivet til de næste i smittekæden. Har man genomsekvenser fra forskellige smittebærere, kan man ved hjælp af matematiske metoder rekonstruere deres genetiske stamtræ. Ved hjælp af stamtræet kan man identificere smittekæder, der kan hjælpe til at forstå virusspredningen. Som vi forklarer senere, kan dette bruges til at forstå effektiviteten af afbødningsstrategier og dermed hjælpe i genåbningsfasen, særligt i forhold til kontaktopsporing.
Fylogenetiske træer
Matematik og genetiske stamtræer
I fagsproget hedder et genetisk stamtræ et fylogenetisk træ, og her findes der en sammenhæng mellem matematikken bag fylogenetiske træer og såkaldte tensornetværk, som er centrale inden for vores felt kvantematematik. Med dette som udgangspunkt satte vi os som mål at bygge det fylogenetiske træ fra de danske coronavirussekvenser samt at identificere smittekæder. Efter to måneders intensivt arbejde har vi offentligtgjort vores resultater på preprintserveren bioRxiv, så det nu er tilgængeligt for forskere og andre interesserede.
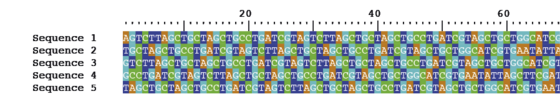
Genomet af den nye coronavirus består af en RNA-sekvens med en længde på cirka 30.000 nukleotider. De fire nukleotider er guanin (G), uracil (U), adenin (A) og cytosin (C). Det er sædvanlig at skrive T i stedet for U, som om det var DNA og ikke RNA. Hvis der sker en mutation, bliver der på en specifik position skiftet en nukleotid ud med en anden, for eksempel blev der i nogle af de danske sekvenser skiftet et A ud med et C i position 15842 (og man skriver så C15842A).
Tensornetværk
Ordne genomsekvenser

Fra man tager en prøve hos en patient, til man har gensekvensen, ligger der et stort arbejde, som bliver udført i Mads Albertsens laboratorium på Aalborg Universitet i samarbejde med Statens Serum Institut, og som blev beskrevet i sidste nummer af Aktuel Naturvidenskab. Det er helt fantastisk, at de sekventerede danske genomer, og også mange andre verden over, bliver lagt offentligt i GISAID-databasen. Dette promoverer deling af virusgenomsekvenser, og det, at vi kunne downloade dem, gjorde vores arbejde muligt.
Genomsekvenserne er næsten ens, men de er forskudt i forhold til hinanden og har måske også andre fejl. Inden man kan undersøge dem, skal man derfor lægge dem præcist ved siden af hinanden (alignment) ved hjælp af nogle computerprogrammer. For at kunne tale om mutationer er det blevet standard i coronaforskningen af bruge en specifik sekvens fra en af de første patienter i Wuhan som reference.
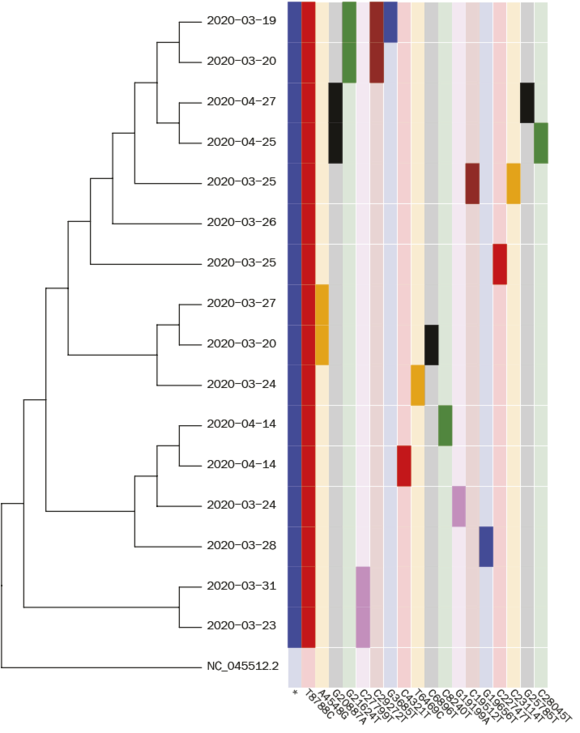
Når det er gjort, kan man bygge et fylogenetisk træ, igen ved hjælp af specielle algoritmer. Vi har også udviklet en speciel visualisering af mutationerne, så vi kan bestemme specifikke smitteveje endnu mere præcist.
Fylogenetisk træ af danske coronavirussekvenser
Smittekæder
Vi er nået frem til, at en stor del (cirka 70 %) af de danske sekvenser har seks specifikke mutationer tilfælles (de seks mutationer tilsammen har navnet A2a2a). Ved at sammenligne dem med data fra Østrig, Norge og Island peger alt på, at en stor del af disse sekvenser stammer fra skisportsstedet Ischgl i Østrig. Det er konsistent med de informationer fra pressemøder, som vi alle sammen husker fra de dage, Danmark lukkede ned. Ved at kigge på flere smittekæder kan vi dog også se andre mindre dele af smitten sandsynligvis kommer fra Italien, Storbritannien og Holland.
Endvidere kan vi se, at virussen allerede i april havde muteret mange gange i Danmark.
Hvis man antager, at én mutation i genomsekvensen på et sted er lige så sandsynlig som en mutation ethvert andet sted, kan man konkludere, at mutationen T8788C er sket først. Dernæst kom mutationen G20887A, som så igen forgrener sig ud i henholdsvis C28045T og G25785T. Vi kan altså direkte aflæse den sandsynlige smittekæde:

Vi har også fundet andre smittekæder, hvor den sidste mutation dukker op i flere sekvenser. Derudover har vi fundet en mutation (C1302T), som internationalt set er mest udbredt i Danmark, men som har spredt sig til blandt andet Island og Sverige. Vi har altså her en smittekæde, som i starten af marts spredte sig fra Danmark, noget som vi ikke før havde hørt beskrevet i medierne.
Et værktøj for fremtiden
Identifikation af disse smittekæder via genomsekventering kan blive vigtigt i fremtiden. Vi ved nemlig, at meget fremtidigt arbejde med at holde epidemien i skak drejer sig om kontaktopsporing. Hvis man bliver testet positiv med coronavirus, skal man have fat i alle, man har været sammen nogle dage inden, så de også kan blive testet. Hvis man nu sekventerer virussen, kan man se, om det kan passe med, at smittekæden rent faktisk er forløbet, som man tror. Hvis ikke, så må kontaktopsporingen udvides.
Udover identifikationen af smittekæder tegner vi et billede af de stammer af virusset, som er i Danmark. Det kan man bruge til at se, om der er en stamme, der får overtaget, hvilket kunne være tilfældet, hvis den var meget mere smitsom. I det data, vi har analyseret, er der ikke noget, der tyder på dette. Derudover kan man bruge det til at se, hvilken indflydelse grænseåbningen og specielt sommerturismen har på de i Danmark forekommende virusvarianter, da andre lande har andre mutationer.
I vores analyse brugte vi det data, der var tilgængelig den 26. maj. I takt med, at pandemien og dens genomdata vokser, kan vi nemt opdatere vores analyse ved at fodre vores computerprogrammer med nye data. Hvis for eksempel specifikke lokale udbrud skal analyseres, kan specielt vores mutationsvisualisering hjælpe med hurtigt at identificere smittekæderne og dermed hjælpe med at imødegå udbruddet.