
Corona-Forskning på speed
Når nøden er størst, er forskningen nærmest. Sådan synes Mads Albertsen fra Aalborg Universitet, at det bør være. Derfor har han og hans forskningsgruppe sat deres bakterieforskning på standby for i stedet at bruge al deres tid på at sekventere arvemasse fra coronavirus.
Af Carsten R. Kjaer, Aktuel Naturvidenskab
Corona-pandemien har sat en kæp i hjulet på utallige forskningsprojekter over hele verden, fordi forskerne er hjemsendt og forment adgang til deres laboratorier. Men samtidig gør den alvorlige situation det også bydende nødvendigt hurtigt at skaffe ny videnskabelig funderet viden om, hvordan vi bedst bekæmper denne trussel mod vores helbred og samfund. Derfor er der stadig hektisk aktivitet i nogle laboratorier, og mange gode kræfter er med kort varsel sat ind på at forske i forskellige aspekter af coronavirussens biologi og effekt på mennesker.
Også forskere, som i udgangspunktet ikke har set sig selv som oplagte “virus-forskere”, har kastet sig ind i kampen mod coronavirussen. Blandt disse er en gruppe forskere på Aalborg Universitet ledet af professor MSO Mads Albertsen. Han og hans forskningsgruppe på 10 personer bruger netop nu al deres tid på at kortlægge coronavirussens arvemateriale i samarbejde med forskere fra Hvidovre Hospital, Statens Serum Institut og Aalborg Universitetshospital. Den information kan forskerne blandt andet bruge til at blive klogere på smittevejene.
Fra bakterier til virus
Normalt fokuserer Mads Albertsens forskningsgruppe ikke på virus, men på bakterier. Da corona-epidemien ramte verden var de i gang med et stort forskningsprojekt kaldet Microflora Danica støttet af Poul Due Jensen Fond, som går ud på at identificere alle bakterier i Danmark. Baggrunden for at kunne gøre det er, at forskerne har opbygget et laboratorium i verdensklasse og ekspertviden om analyse og identifikation af bakterier.
»Metoderne til at identificere bakterier bygger på sekventering og analyse af deres arvemasse«, fortæller Mads Albertsen.
Sekventering betyder konkret, at man kortlægger rækkefølgen af de byggeklodser (nukleotider), som arvemassen består af. Mads Albertsen fortæller, at der principielt ikke er forskel på metoderne til at sekventere arvemateriale fra bakterier og virus. Bakteriers arvemasse er i form af DNA, som består af to strenge snoet om hinanden, mens coronavirus’ arvemasse er i form af RNA, som kun består af en enkelt streng.
»I praksis betyder det blot, at vi skal tilføje et enkelt ekstra trin i proceduren, hvor vi laver virus-RNA’et om til DNA ved at tilsætte nogle specifikke reagenser og varme det hele op. Derudover foregår alt præcis på samme måde. Det har betydet, at vi har kunnet stille hele vores pipeline til rådighed for sekventering af arvemateriale fra coronavirus på meget kort tid,« fortæller Mads Albertsen.
Hjælp til et presset sundhedssystem
Moderne apparater til sekventering, som dem Mads Albertsen og kolleger bruger i deres laboratorium, fylder ikke mere end man kan have det i lommen, og sekventering af virus-DNA er også noget, der allerede rutinemæssigt foretages lokalt på hospitalerne. Når Mads Albertsen og hans forskningskolleger stiller sig til rådighed i kampen mod coronavirus handler det derfor først og fremmest om at løse et akut kapacitetsproblem.
»Lokalt kan en enkelt person med et sekventeringsapparat måske kortlægge 25 virusgenomer om dagen, hvis vedkommende bruger al sin tid på det. Vi har til sammenligning i vores laboratorium kapacitet til at generere flere hundrede virusgenomer om dagen, så da det blev klart for os, at der var et akut behov for at sekventere virus-DNA, var vi ikke et øjeblik i tvivl om, at det eneste rigtige var at byde os til. Sundhedspersonalet har jo så rigeligt at lave for tiden, så hvis vi kan være med til at tage noget af presset, gør vi det med glæde,« siger Mads Albertsen.
Genomer i hobetal
I løbet af kun et par uger lykkedes det Mads Albertsen af få alt på plads sammen med sine samarbejdspartnere, og de første 600 virusgenomer er i skrivende stund på vej ud af pipelinen. Analyse af de mange genomer kan nu være med til at afklare, hvordan smitten har spredt sig i Danmark. Når det er relevant at kigge på virusgenomer i den sammenhæng er det fordi, virus’ arvematerialer ændrer sig meget hurtigt på grund af mutationer. Derfor kan man ved hjælp af analyser af variationer i coronavirussens arvemasse opstille en slags stamtræ for virussen, der kan bruges til at kortlægge smittevejene.
I flere andre lande er der blevet etableret lignende forskningskonsortier, som genererer virusgenomer i stort antal. De danske genomer kommer derfor til at indgå i de verdensomspændende databaser, så forskere over hele verden kan samarbejde om at forstå og bekæmpe den globale coronavirus-pandemi.
»Faktisk er det ikke det rent tekniske i omstillingen til at sekventere virus-arvemasse, der er udfordringen i et sådant projekt,« fortæller Mads Albertsen. »Det tekniske kan i princippet klares på en eftermiddag – det er alt det “udenom”, der kræver en særlig indsats.«
Den største flaskehals i sekventeringsarbejdet er computerkraft, idet det kræver masser af beregningskraft at processere de data, der kommer ud af sekventeringsmaskinerne. Her var forskerne afhængige af, at et team af universitetets IT-folk kunne smide, hvad de ellers havde i hænderne for at bygge en server dedikeret til projektet. Derudover var der også en juridisk hurdle i at skulle omstille sig til at arbejde med prøver fra patienter i stedet for prøver af for eksempel spildevand med bakterier. Men også alle spørgsmålene om, hvordan de personfølsomme data skulle håndteres blev afklaret i rekordfart med assistance fra universitetets jurister.
Om coronavirus
Coronavirus har været kendt siden 1930’erne, og der kendes flere end 30 forskellige stammer af coronavirus. Kun 7 af disse er kendt for at kunne fremkalde sygdom hos mennesker. Men coronavirus kan mutere og på den måde pludselig få evnen til at inficere mennesker, selvom den før kun har cirkuleret i dyr. Det er således dette, der menes at være sket med den aktuelle coronavirus – med det borgerlige navn SARS-CoV-2, der forårsager sygdommen Covid-19.
SARS-CoV-2 er en stor virus, og dens arvemasse (genomet) består af RNA opbygget af knap 30.000 nukleotider. Forskere har fundet, at denne arvemasse indeholder instruktioner til op mod 29 forskellige proteiner, der sørger for alt fra at lave kopier af coronavirussen til at undertrykke værtens immunsystem.
Den genetiske kode ændrer sig dog ret hurtigt i coronavirus på grund af en forholdsvis høj mutationsrate. Faktisk viser en analyse af virus fra 600 danske patienter, som virusforsker Anders Fomsgaard fra Statens Serum Institut har lavet sammen med Mads Albertsen, at den genetiske kode hos virussen hele tiden ændrer sig en lille smule, således at virus hos den enkelte patient har en unik genetisk profil.
Langt de fleste mutationer hos virus har ingen betydning for hverken virussen selv eller de mennesker, den inficerer. Potentielt kan en mutation dog gøre virussen mere dødbringende, men det kan selvfølgelig også gå den anden vej – og det sidste er nok det mest sandsynlige.
Det er således vigtigt at følge med i, hvilke nye mutationer, der dukker op, da de kan have betydning for sygdomsudviklingen. Specielt er det interessant at studere mutationer, som er fælles og begynder at dominere i viruspopulationen, da det vil tyde på, at netop disse mutationer har givet virus en evolutionær fordel.
Sammenligninger af virusgenomer kan også hjælpe med at afsløre smittevejene. Når virus introduceres i et samfund opstår der lokale “smitteklynger”, hvor virussen breder sig hurtigt i en lokal gruppe af mennesker fra en enkelt inficeret patient. Virus fra patienter i en smitteklynge vil have en tendens til at dele de samme mutationer, da de alle et nært beslægtede med virussen fra den første patient i smitteklyngen. Det giver mulighed for at se, hvorfra virus er kommet og at kortlægge smitteklyngernes spredning i verden på baggrund af en sammenligning af, hvilke mutationer virus fra forskellige patienter har tilfælles.
Når systemet virkelig rykker
»Jeg har i det hele taget haft en oplevelse af, at hver gang jeg havde et problem, så stod der et helt team af specialister klar til at hjælpe mig med at få det løst,« fortæller Mads Albertsen. På den måde har hans engagement i corona-forskningen først og fremmest givet ham den positive erfaring, at når det virkelig gælder, kan det danske forskningssystem rykke helt utroligt hurtigt. Med til historien hører således også, at forudsætningen for, at Mads Albertsen og kolleger kunne gå i gang med at sekventere virus-arvemasse var, at Poul Due Jensen Fonden med dags varsel gav tilladelse til, at hele teamet af forskere i Microflora Danica-projektet, kunne bruge al deres tid på coronavirus i stedet.
Rent fagligt forventer Mads Albertsen til gengæld ikke, at han vil lære en masse han kan bruge i sit fremadrettede arbejde.
»Vi har jo egentlig bare gjort det, vi allerede gjorde i forvejen,« siger han. »Vi er glade for at kunne hjælpe, men jeg regner ikke med, at vi fremover kommer til at beskæftige os noget videre med virus. Lige den del, der handler om at håndtere personfølsomme oplysninger, kan jeg dog godt forestille mig, at vi kan få glæde af på længere sigt, « siger han.
På det personlige plan er der måske også et efterslæb på hjemmefronten, han skal have rettet op på, når situationen igen er normaliseret. Arbejdet med corona-projektet fra morgen til aften foregår fra et interimistisk hjemmekontor i forældrenes stue, da Mads Albertsen faktisk ikke selv er i laboratoriet. Og således er al husarbejdet og pasningen af tre børn derhjemme i alderen 0 til 6 for tiden overladt til kæresten.
»Heldigvis er hun også glad for, at jeg kan bidrage, men jeg kommer nok til at støvsuge og skifte ble de næste par år...,” slutter Mads Albertsen.
Sekventering
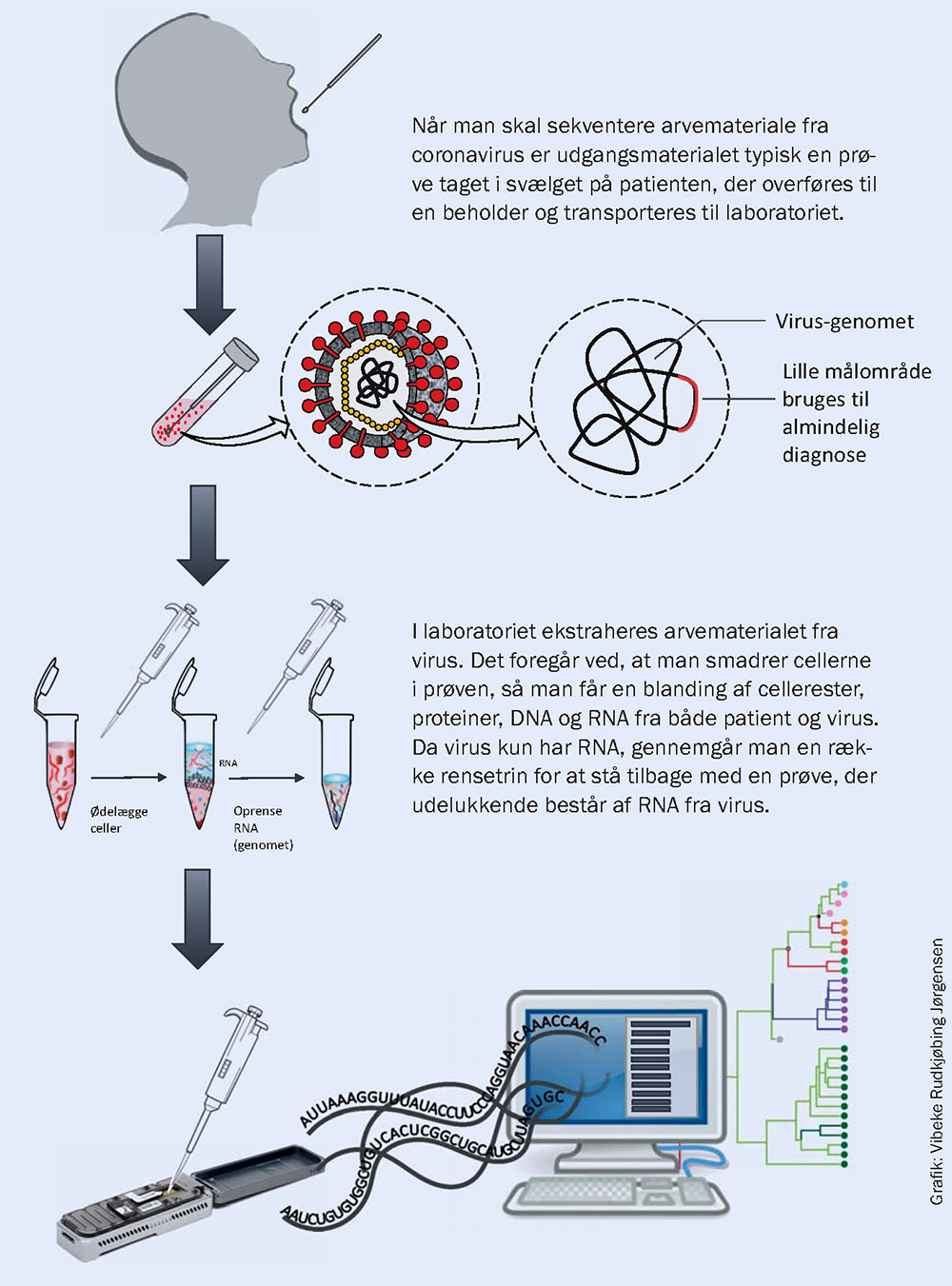
Efterfølgende sekventerer man virus-RNA’et i prøven (dvs. bestemmer rækkefølgen af nukleotider). Det foregår ved hjælp af en lille, kompakt sekventeringsmaskine (en såkaldt nanopore). Med en del computerarbejde kan man derefter sammenstykke aflæsningerne af de mange stykker RNA til et komplet genom for virussen. Ved den normale diagnose analyseres der kun for en lille del af genomet, og resultatet afslører ikke sekvensen, men måles ved hjælp af farveintensitet, da dette sparer tid og penge i forhold til genom-sekventering.
Når man har tilpas mange genomer er det muligt at lave såkaldte (fylogenetiske) stamtræer, hvor man grupperer genomerne efter, hvor meget de ligner hinanden (ud fra mutationerne). Det har vist sig at kunne bruges til at se, hvor virus i de forskellige danske patienter er kommet fra. På figuren kunne de grønne grene på træet eksempelvis være blevet smittet i Østrig og de lilla i Italien. I takt med, at virus muterer og smitte foregår indenfor Danmark, vil vi efterhånden begynde at se nogle nye grupperinger af grene, hvor genomet er forskellig fra dem, der ses hos danskere, der blev smittet i udlandet og også forskellig fra dem, der findes i andre lande. Hvis man kobler disse oplysninger med patientinformation, vil man kunne opspore potentielle hotspots for smitte.

