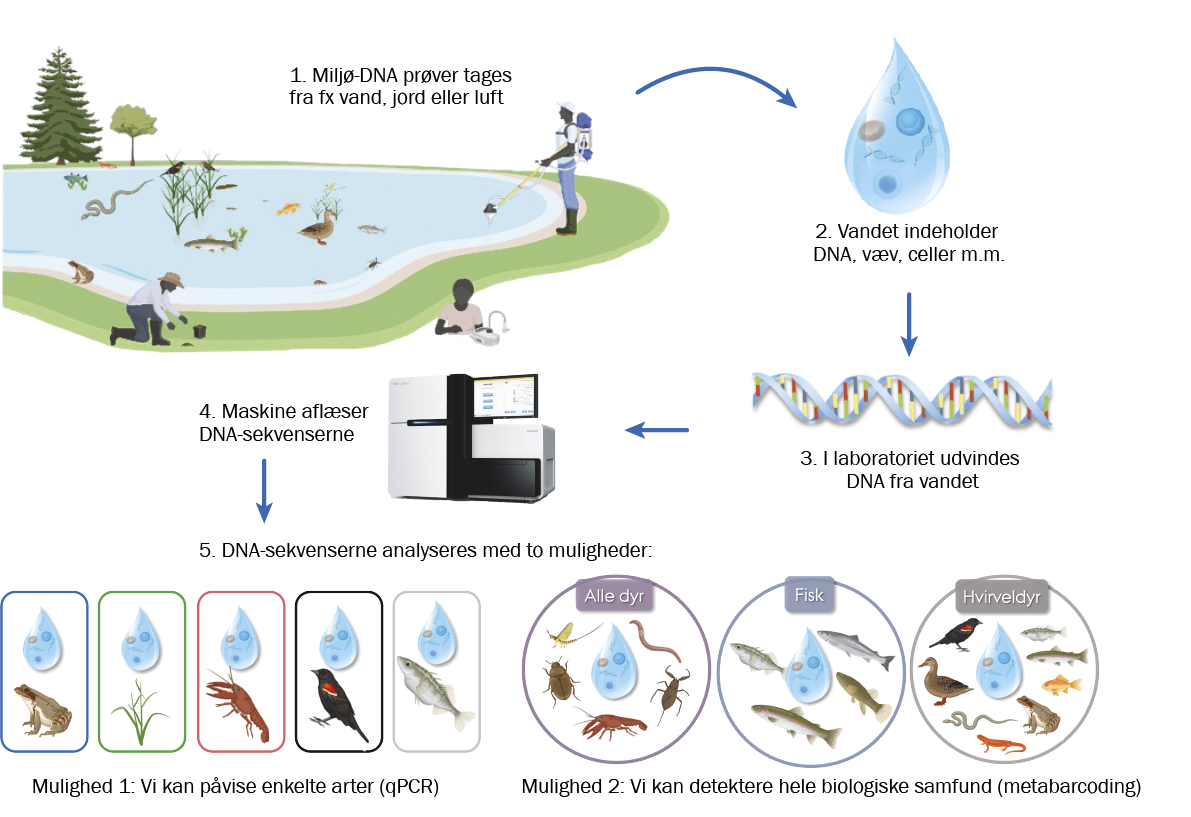
Miljø-DNA kan revolutionere naturovervågningen
Gennem de seneste to årtier har forskere revolutioneret mulighederne for naturovervågningen gennem analyser af miljø-DNA. Danske forskere har været pionerer på de metoder, der benyttes til at analysere DNA i naturen. Og de metoder tillader dem både at overvåge nutidens natur samt studere verden, som den så ud engang for mange tusinder af år siden.
Af Kristian Sjøgren
Allerede i 1980’erne opdagede forskere, at analyser af DNA i en miljøprøve kunne afsløre mange flere mikroorganismer end den traditionelle metode, hvor man først skulle kultivere bakterier på agarplader for at finde ud af, hvad der var i prøven. DNA-analyserne viste også mange typer af bakterier, som man ikke havde set før.
Disse første analyser af DNA i jordprøver var startskuddet til det, der i dag kendes som analyser af miljø-DNA (også kaldet eDNA = environmental DNA). Det er et forskningsfelt, man ikke kan omtale uden samtidig at nævne Danmark, idet danske forskere har været pionerer indenfor området og udviklet mange af de metoder, der i dag benyttes til blandt andet at overvåge fiskebestande i alverdens have og undersøge, hvad der levede på Jorden for tusinder af år siden.
En af de forskere, som har været med et godt stykke af vejen, er lektor Philip Francis Thomsen fra Institut for Biologi ved Aarhus Universitet, og han fortæller, hvordan det hele startede.
»Efter de første fund af DNA i miljøprøver gik der nogle år, hvor der ikke skete så meget udvikling indenfor feltet, men i 2003 begyndte det virkelig at tage fart, da Eske Willerslev fra Center for Geogenetik ved Københavns Universitet gik ind i feltet og begyndte at undersøge, hvad det rent faktisk var muligt at sige ud fra prøver af is og permafrost,« forklarer Philip Francis Thomsen.
Philip Francis Thomsen var selv ph.d.-studerende under Eske Willerslev og har som sådan været med næsten helt fra begyndelsen.
Forskellige teknikker til at analysere miljø-DNA
Analyser af miljø-DNA kan udføres på forskellige måder afhængigt af formålet med analysen. Den grundlæggende forskel ligger i, om man ønsker at identificere specifikke arter eller opnå en bred indsigt i alt DNA i en given prøve.
Skal man lede efter specifikke arter, benytter forskere målrettede metoder såsom PCR (Polymerase Chain Reaction). Ved PCR mangfoldiggøres DNA-sekvenser, der er unikke for den ønskede art, og det sker ved hjælp af specifikke “primere”, der binder til DNA’et. Denne metode er særdeles følsom og gør det muligt at identificere selv små mængder DNA fra den art, som man undersøger en prøve for tilstedeværelsen i. Metoden er især nyttigt, hvis man har en idé om, hvilke arter man leder efter, eller når prøven indeholder mange forskellige DNA-fragmenter, som kan skabe “støj”. En variant af denne metode er qPCR, som både kan kvantificere DNA'et og verificere dets tilstedeværelse.
Ved brede analyser af DNA i en prøve anvender forskere i stedet metabarkodning eller shotgun-sekventering. Metabarkodning bruger universelle primere til at mangfoldiggøre DNA fra en lang række organismer i prøven. Herefter bestemmer forskerne rækkefølgen (sekvensen) af DNA-byggesten i de mangfoldiggjorte DNA-fragmenter og bestemmer deres oprindelse ved at matche dem mod enorme databaser med DNA-sekvenser fra alle tænkelige organismer, der nogensinde er blevet bestemt. Shotgun-sekventering går skridtet videre og sekventerer alt DNA i prøven uden en forudgående proces, hvor DNA’et mangfoldiggøres. Denne metode giver en bred og detaljeret indsigt i den samlede biodiversitet, men kræver store mængder data og ressourcer at analysere. Valget af metode afhænger således af målet med analysen, prøvens kvalitet og de tekniske ressourcer til rådighed.
Miljø-DNA i havvand
Mens man til undersøgelser af miljø-DNA i en sø kan klare sig med prøvestørrelser svarende til et snapseglas, skal der større mængder til – svarende til et ølglas – i saltvand for at få DNA nok. Figuren viser, hvordan Philip Francis Thomsen og kolleger ved at tage prøver tre forskellige steder omkring Helsingør havn kunne finde DNA fra mange af de almindelige fisk, man umiddelbart ville forvente at finde. En overraskelse var tilstedeværelsen af DNA fra sardin – men det var blot et forvarsel om, at denne fisk faktisk allerede var ved at etablere sig i Danmark. Figur efter Thomsen et al. (2012b).
Figuren viser, hvor mange arter af fisk, forskellige indsamlingsmetoder kunne påvise i Helsingør. Det fremgår, at miljø-DNA er den metode, der opdager flest arter. De to søjler med miljø-DNA er den samme prøve analyseret med fem års mellemrum og viser, at metoden i mellemtiden var blevet kraftigt forbedret (ikke at der var kommet flere arter til!). Figur efter Valentini et al. (2016).
Kortlagde faunaen i Sibirien for titusinder af år siden
Det var tilbage i 2003, at Eske Willerslev med sin forskningsgruppe fik verdens øjne op for, hvad det er muligt at udtale sig om med en lille klump jord eller en vandprøve. Forskerne lavede dengang et banebrydende forsøg, der blev omtalt i hele verden og viste, at det ud fra en lille sedimentprøve fra den sibiriske permafrost var muligt at genfinde DNA fra mange af de dyr, som levede i Sibirien for titusinder af år siden.
DNA i naturen stammer fra hud, hår eller andre celler, som dyr hele tiden afgiver til omgivelserne. Det gør vi mennesker for den sags skyld også. DNA’et vedbliver dog ikke med at være i naturen, men nedbrydes over tid, og den proces er meget afhængig af temperatur. Netop i permafrosten blev DNA’et bevaret i tusinder af år, og forskerne kunne derfor finde DNA fra mammutter, bisoner, lemminger, planter og meget mere. Det var med andre ord muligt at genskabe et billede af floraen og faunaen, som det alt sammen så ud i en meget fjern fortid. Miljø-DNA gav på den måde en indsigt, som det ville have taget forskere årtier at komme frem til, hvis de skulle have analyseret knoglestumper og frosne frø og nødder.
»Det var chokerende, så meget man kunne se ud fra analyser af jordprøver. Det var virkelig der, at der blev skabt en helt ny æra indenfor miljøbiologi, hvor mange fik øjnene op for, hvad det egentlig er muligt at gøre med denne type analyser af alt DNA i en prøve,« forklarer Philip Francis Thomsen.
Nye metoder revolutionerede naturovervågning
Der gik ikke mange år fra det danske gennembrud, før forskere over hele verden begyndte at tage teknologien til sig og lave deres egne forsøg. Blandt andet benyttede franske forskere miljø-DNA til at lede efter invasive amerikanske oksefrøer i franske søer. De franske forskere viste med deres studie, at miljø-DNA ikke bare er relevant i undersøgelser af fortidens DNA, men at det også kan være meget relevant i undersøgelser af nutidens økologi. Med det franske studie kunne forskerne pege på, hvilke franske søer der viste tilstedeværelse af de invasive oksefrøer, og hvilke der indtil da var gået fri.
Philip Francis Thomsen blev ansat som ph.d.-studerende ved Center for Geogenetik under Eske Willerslev kort efter, at det franske resultat var publiceret, og i sin ph.d.-afhandling bragte Philip og hans kollegaer de teknikker, som forskerne benyttede i deres analyser, skridtet videre.
Indtil da havde forskere undersøgt miljøprøver for specifikt DNA i prøverne. Det vil sige, at de i deres undersøgelser måske ville kigge efter oksefrøer og så lede specifikt efter oksefrø-DNA i prøverne. I Philip Francis Thomsens ph.d.-afhandling forsøgte forskerne dog noget helt nyt, og det var at tjekke for alle arter samtidig. Det lader sig gøre med teknikker som metabarkodning og shotgun-sekventering (se faktaboks). Det vigtigste er at forstå, at det med de nye teknikker ikke er nødvendigt at vide, hvad man leder efter i prøverne. Man kan bare lede.
I sine forsøg viste Philip Francis Thomsen blandt andet, at det er muligt at kortlægge alle padder og fisk i en hel sø blot ved at analysere nogle vandprøver fra søen. Så kunne forskerne sige, om der var gedder, aborrer eller forskellige arter af frøer i søen. Fors kerne gjorde det samme med vandprøver fra havet omkring Helsingør, hvor de kunne identificere tilstedeværelsen af blandt andet sild, torsk, rødspætte og skrubbe. I et andet studie fra Danmark viste forskerne, at de også kunne finde DNA fra marsvin i prøverne, og at der derfor havde været marsvin i hav området på et tidspunkt forinden. Forskerne lavede også et lignende forsøg i dybhavet ud for Grønland og kunne vise, at det DNA, som forskerne identificerede i vandprøverne fra dybt nede i havet, matchede det, som fiskerne hev op med deres trawl.
»Det var her, at det virkelig blev klart, hvordan man kunne bruge analyser af miljø-DNA i miljøovervågning. Da jeg startede på min ph.d., var vi nok omkring fem forskningsgrupper i verden, der arbejdede med miljø-DNA, og i dag er der mange hundrede grupper. Det vidner noget om, hvor store perspektiver folk ser i teknologien,« siger Philip Francis Thomsen.
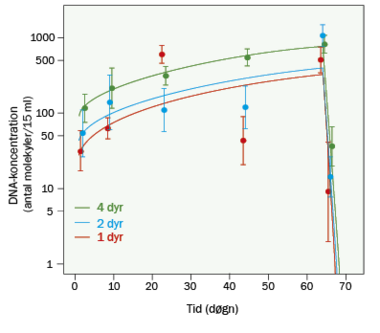

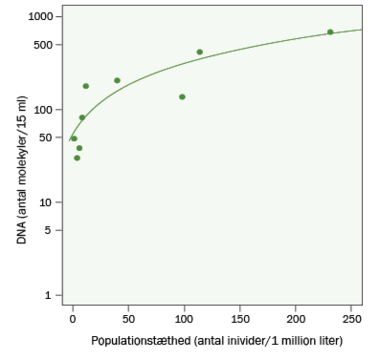
Mængden af DNA matcher antallet af dyr
Det næste store fremskridt indenfor forskningen var påvisningen af, at det ikke kun er muligt at identificere arter ud fra miljø-DNA, men at det også er muligt at kvantificere dem. Philip Francis Thomsens forskning viste blandt andet, at der er en sammenhæng mellem mængden af DNA fra det enkelte dyr i en prøve og så det faktiske antal dyr i miljøet. Det viste forskerne blandt andet ved at analysere vandprøver fra søer med forskellige tætheder af padder og supplere disse prøver med kontrollerede akvarieforsøg med haletudser. Denne forskning viste også, i hvor lang tid DNA egentlig forbliver i vandmiljøet. Resultaterne viste, at DNA-signalet typisk forsvinder efter én til to uger efter, at dyrene er borte.
»Det viste, at hvis vi ser et signal fra et dyr i en vandprøve, har dyret været der indenfor relativ kort tid. Det var vigtigt at få slået fast, da mange troede, at det DNA, som vi fandt, bare var blevet ophobet over tid og dermed ikke kunne sige noget om, hvad der lever i miljøet lige nu og her. Vi viste også, at vi ud fra mængden af DNA kan sige noget om antallet af dyr. Begge dele er meget relevante at vide ved naturovervågning, hvor man jo netop gerne vil kortlægge, om der er givne dyr i et område, og eventuelt hvor mange der er,« siger Philip Francis Thomsen.
Hvalhajer i Qatar
Efter han var færdig med sin ph.d. og var blevet ansat som postdoc, tog Philip Francis Thomsens forskning ham til Qatar, hvor han med sine kollegaer og samarbejdspartnere satte sig for at undersøge tilstedeværelsen af hvalhajer omkring nogle boreplatforme. Der var dog ingen tvivl om, at hvalhajerne var i miljøet. Det kunne man se med det blotte øje; det er trods alt verdens største fisk. I stedet var forskerne interesserede i, om de ud fra miljø-DNA ikke bare kunne identificere tilstedeværelsen af hvalhajerne, men oveni pege på, hvilke bestande det drejede sig om.
Hvalhajer, ligesom alle andre dyr, har ikke bare gener, der adskiller dem fra andre arter, men også genvariationer, der adskiller grupper af individer fra hinanden. Indenfor biologien hedder det haplotyper. Forskerne havde til projektet adgang til vævsprøver fra hvalhajerne og kunne i dem se fordelingen af haplotyper hos de hvalhajer, der svømmede rundt omkring boreplatformene ved Qatar. Da de undersøgte miljø-DNA for de samme genetiske markører, fandt de den samme fordeling. Det viste, at forskerne med miljø-DNA ikke bare kunne vise, at der var hvalhajer i vandet, men også hvilken genetisk sammensætning denne population havde.
»Vi kan endnu ikke skelne de enkelte individer fra hinanden, men med undersøgelser af haplotyper fra vandprøverne er det muligt at sige noget om den genetiske variation, som findes i en population. Det gav nogle helt nye muligheder indenfor naturovervågning,« forklarer Philip Francis Thomsen.

Kortlægning af fisk ved kysterne i Danmark
I Qatar lavede forskerne også analyser af miljø-DNA fra koralrev, sandbanker, mangrover og alt muligt andet og viste derigennem, at det med miljø-DNA er muligt at overvåge et helt lands artsdiversitet af marine hvirveldyr som for eksempel fisk. Disse undersøgelser blev inspirationen til den forskning, som Philip Francis Thomsen kastede sig over, da han kom tilbage til Danmark, hvor han også flyttede til Aarhus Universitet og startede sin egen forskningsgruppe. Den næste række af undersøgelser viste, at det ud fra over 100 vandprøver fra kystområder i Danmark var muligt at kortlægge store dele af kongerigets fisk. Fordi forskerne havde adgang til så mange prøver, var det muligt for dem at lave et digitalt Danmarkskort, hvor man kunne se fordelingen af alle de fisk, som lever i havet omkring os. Her identificerede forskerne DNA fra 52 kystnære fiskearter, hvilket er omkring 80 % af alle de kystnære fisk, der nogensinde er observeret i kystnære biotoper. Forskningen blev lavet i samarbejde med 360 frivillige fra Danmarks Naturfredningsforening.
Philip Francis Thomsen fortæller, at med kortlægningen er miljø-DNA blevet et uvurderligt redskab til overvågning af havmiljøet omkring Danmark. For eksempel viste analyserne af miljøprøverne meget tydeligt, hvor den sortmundede kutling lever i Danmark. Den sortmundede kutling er en invasiv art, og det er derfor også meget relevant hele tiden at følge dens udbredelse. Med analyserne viste forskerne, at på analysetidspunktet levede fisken kun i havet omkring Bornholm og omkring Sjælland, men endnu ikke rundt om Jylland. Analyser af miljø-DNA kan over de kommende år vise, om udbredelsen ændrer sig, og om den sortmundede kutling kommer yderligere vestpå, eller om den holder sig til de områder, hvor den lever i dag.
»Rundt om i verden benytter mange lande miljø-DNA til at overvåge forskellige relevante arter. Det kan være invasive arter som den sortmundede kutling eller amerikansk oksefrø, eller det kan være truede dyrearter som for eksempel stor vandsalamander. Det betyder også, at der er blevet etableret mange firmaer, som har specialiseret sig i analyser af miljø-DNA, og som de udfører for myndigheder eller NGO’er,« siger Philip Francis Thomsen.
Forskeren advarer dog, at man ikke bør forkaste alle de måder, man hidtil har lavet naturovervågning på for at erstatte dem med analyser af miljø-DNA.
»Der kan være problemer i forhold til at skifte metode, fordi man derved bryder lange tidsserier med data op. Har man som eksempel indsamlet insekter og talt dem i 30 år, skal man blive ved med at gøre det, fordi det giver nogle helt unikke tidsserier, som man endnu ikke kan skabe med miljø-DNA, før metoden har været brugt i mange år. Netop tidsserier er noget af det vigtigste at have adgang til indenfor forskning i, hvordan det går naturen over tid,« siger Philip Francis Thomsen.
Analyserede DNA fra insekter på blomster
Netop når det kommer til insekterne, er de også i centrum i et af de seneste af Philip Francis Thomsens forskningsprojekter. I det projekt ønskede han at komme dybere ned i en af de store bekymringer for biodiversiteten lige nu og her, og det er den udbredte insektdød, som rapporteres fra mange studier i disse år.
I forskningsarbejdet indsamlede han derfor blomster og analyserede alt det DNA, som fandtes på blomsterne. Helt specifikt kiggede han efter DNA fra insekter. Analyserne afslørede, hvor overvældende naturen nogle gange kan være, idet forskerne samlet set identificerede DNA fra intet mindre end 135 insektarter fra bier og biller til fluer og sommerfugle. Der var både bestøvere, rovinsekter og dem, der blot lever af at spise pollen. Der var også insekter, som lever af at lægge æg i andre insekter, for eksempel en art af snyltehveps, som parasiterer en art af bladlus. I nogle af prøverne fandt forskerne endda både DNA fra disse parasitiske insekter og den vært, som de lægger deres æg i. Miljø-DNA gav på den måde en indsigt i insekternes verden, som er meget svær at få på anden måde.
»Når vi taler om insekters tilbagegang, kan man tage en blomst og så undersøge DNA for at se, hvilke insekter der har været på blomsten indenfor den seneste tid. Dette er vanskeligt at kortlægge ved at sidde og studere en blomst. For det første er det ikke praktisk muligt, og for det andet er det for mange af disse arters vedkommende svært at skelne dem fra hinanden med det blotte øje. Det kan man til gengæld let gøre ved at se på deres DNA,« siger Philip Francis Thomsen, der forudsiger, at de næste store skridt indenfor brugen af miljø-DNA kommer til at omhandle netop undersøgelser af prøver fra land fremfor vand.
»Det er et spørgsmål om tid, før det her også bliver lige så stort indenfor naturovervågning, som undersøgelser af vandprøver er det. Det er jeg overbevist om,« siger han. ♦