Genetisk resistens mod infektionssygdomme
En ny metode til at typebestemme DNA har vist sig særdeles velegnet til at identificere fisk med naturlige anlæg for sygdomsresistens. Metoden giver forskerne mulighed for at lokalisere gener, der koder for naturlig og medfødt modstandskraft mod diverse infektionssygdomme – potentielt også i mennesker.
Af Kurt Buchmann, Per W. Kania, Asma M. Karami og Heidi Mathiessen
I en pandemi-tid med en høj risiko for at blive ramt af en potentielt dødelig infektionssygdom diskuterer vi naturligvis hyppigt, for ikke at sige dagligt, hvilke personer og befolkningsgrupper, som er mest udsat for at udvikle sygdom. Epidemiologisk forskning kan give nogle solide fingerpeg om, hvilke gener, der hos mennesker, giver enten modtagelighed eller resistens mod en given mikroorganisme, uanset om vi snakker om virus, bakterier eller parasitter. Det er dog velkendt, at kontrollerede dyreforsøg kan give os endnu mere indsigt i mekanismerne, og brugen af fisk som forsøgsdyr har nu vist sig at åbne muligheder for at give en langt mere præcis viden på området.
Fisk har i mange sammenhænge vist sig at fungere som en eminent forsøgsdyrmodel, idet man har mulighed for at bruge klart genetisk definerede dyr, som er blevet skabt og opdrættet under præcis de samme forhold. Resultaterne påvirkes således ikke af andre sygdomme, fødeadgang, kønsforskelle og fysisk-kemiske forhold i øvrigt. Der er således ikke så mange andre faktorer, som kan påvirke forsøgsresultatet, når man ønsker at belyse betydningen af en enkelt faktor. Desuden kan man anvende et betydeligt antal dyr, som giver basis for en solid statistisk analyse. I vores forskning ved Københavns Universitet anvender vi i vid udstrækning regnbueørreden Oncorhynchus mykiss som model.
Typebestemmelse af DNA
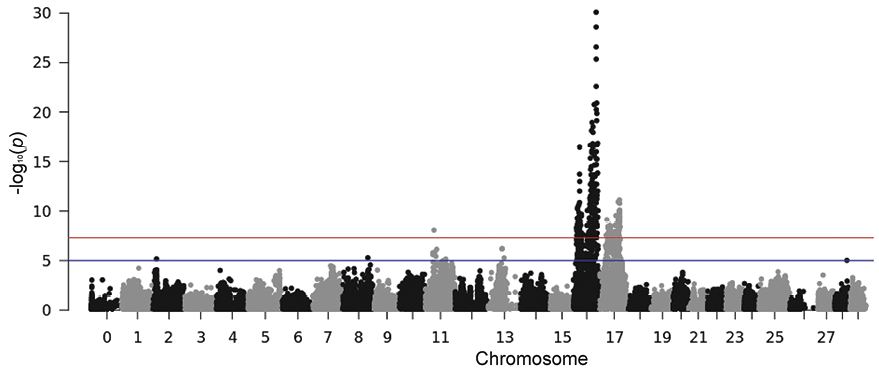
Vi har for nylig offentliggjort to studier, der viser, at gener, der koder for sygdomsresistens mod en frygtet parasit og en nok så kendt bakterie, er præcist lokaliseret på ørredens kromosomer (henholdsvis kromosom nummer 16/17 og 21). Vi har i studierne anvendt en ny teknik til at typeinddele DNA ud fra identificering af specifikke områder i DNA’et, hvor gensekvensen kan bære en udskiftning af et enkelt nukleotid med et andet – for eksempel kan et A (adenin) i en sekvens være udskiftet med T (thymin). Dette kaldes på fagsproget enkeltnukleotidpolymorfi eller blot SNP efter det engelske Single Nucleotide Polymorphism. Metoden kan i hver enkelt fisk detektere 57.501 specifikke enkeltnukleotidpolymorfier på ørredens 29 kromosomer.
Et grundigt forarbejde har tillige kortlagt placeringen af de pågældende enkeltnukleotidpolymorfier på kromosomerne, og da regnbueørredens genom i vid udstrækning er kortlagt, giver det os nu mulighed for at give gode bud på hvilke gener, som er involveret i resistens over for sygdommene. Sammensætningen af enkeltnukleotidpolymorfier varierer fra individ til individ. Når man karakteriserer et individ ved brug af mere end 50.000 af disse genområder, kan man se forskel på individer med forskellige anlæg for sygdom.
Placeringen af 57.701 enkeltnukleotidpolymorfier (SNP-markører) på regnbueørredens 29 kromosomer er kortlagt. Derved kan man finde de kromosomer, der bærer gener for naturlig modstandskraft. Her ses kromosomerne nr. 16 og 17 at være forbundet med beskyttelse mod fiskedræberparasitten Ichthyophthirius multifiliis. Værdien på Y-aksen angiver sandsynligheden for, at den enkelte SNP er repræsenteret i de modstandsdygtige regnbueørreder.
Parasitter og bakterier
Vi har set nærmere på bakteriesygdommen vibriose, der forårsages af bakterien Vibrio anguillarum. Den bakterie er beslægtet med Vibrio cholerae, som fremkalder kolera hos mennesker. Et angreb i en fiskepopulation vil som oftest slå mere end halvdelen af fiskene ihjel. Man kan inficere fiskene eksperimentelt blot ved at tilsætte en kultur af de sygdomsfremkaldende bakterier til en fisketank med fisk. Parasitsygdommen, som vi har sat under lup, er hvidpletsyge, på latin benævnt Ichthyophthirius multifiliis, som ubehandlet kan udrydde hovedparten af en hel population af fisk. Derfor kaldes den også for fiskedræberen.
Det var slående, at gener, der koder for ørredens modstandskraft over for parasitten og bakterien sidder på forskellige kromosomer. På kromosom nr. 21 sidder generne, der giver ørreden en naturlig resistens over for bakterien Vibrio anguillarum. Derimod sidder generne, som giver fisk modstandskraft mod fiskedræber-parasitten, på kromosom nr. 16 og 17 (se figur). Noget tyder altså på, at vidt forskellige mekanismer er i brug.

Hvilke gener bliver aktiveret?
I forbindelse med de eksperimentelle infektionsforsøg, som er gennemført på Københavns Universitet, blev der ikke alene udtaget prøver af alle fiskene til typebestemmelse af deres DNA. Der blev også taget RNA-prøver af henholdsvis syge og overlevende fisk for at udføre såkaldte gen-ekspressions-analyser, som viser, hvilke gener i immunsystemet der er aktive på et givet tidspunkt. Man kan med den metode sammenligne fisk, der bliver syge, med de fisk, som forbliver raske og overlever. Det var således tydeligt, at en række gener, som indgår i det medfødte (innate) immunsystem, blev aktiveret af de invaderende sygdomskim.
Derudover viste det sig, at også gener i det såkaldt tillærte (adaptive) immunsystem blev opreguleret meget tidligt efter mødet med mikroorganismerne. Det gjaldt især nogle særlige typer antistoffer (IgD), som meget hurtigt indgik i fiskens reaktion tidligt, efter smitten blev givet. Regnbueørredens kromosom nr. 21, som var afgørende for fiskens modstandskraft mod Vibrio-bakterien, huser netop en lang række gener for antistoffer. Det giver et fingerpeg om, i hvilken retning vi skal arbejde i fremtiden.
Bedre forståelse af sygdom og død
Forsøgene blev udført med regnbueørreder som forsøgsdyr. De var omhyggeligt opdrættet under helt sygdomsfri forhold, indtil vi eksponerede dem for en veldefineret sygdom. For at sikre, at en meget bred vifte af gener var repræsenteret i studiet, havde vi desuden produceret et stort antal familier baseret på et stort antal forældrefisk, som alle var genetisk veldefinerede. Resultaterne kan anvendes på mange områder. Der er et væsentligt element af basal videnskab i studiet, idet vi med de nye metoder har fået en mulighed for at sætte fokus på gener i værtsdyret, som måske kan forklare tilsvarende sammenhænge også i højere dyr, herunder mennesket. Studiet kan i hvert fald hos fisk besvare spørgsmålet: Hvem bliver syg, og hvem overlever? Og det er meget aktuelt i en verden, hvor man opdrætter fisk for at føde befolkningen.
Mulighed for bedre fiskevelfærd
Kendskabet til sammenhængen mellem enkeltnukleotidpolymorfier og sygdomsresistens giver mulighed for meget hurtigt at avle en ny generation af fisk, der har anlæg for en høj modstandskraft. Det er naturligvis en efterspurgt vare overalt i verden, hvor sundhed og fiskevelfærd står højt på ønskelisten. Man anvender således betegnelsen Quantitative Trait Loci (QTL) for denne viden om gener i et avlsdyr, som giver opdrætteren mulighed for at producere nye generationer af dyr med de ønskede egenskaber.
Danske producenter af ørredæg vil med den ny viden være interessante på det internationale marked, som hvert år efterspørger 3 milliarder æg til fiskeopdræt. Traditionel avl er baseret på, at man lader overlevende dyr vokse op og kønsmodnes, hvorefter man kan avle videre på dem. Det tager dog mange år, idet generationstiden for fisk som regnbueørreden kan være flere år. Med de nye redskaber i hånden kan man straks, når analyseresultatet foreligger, gå i gang med at producere en ny generation med en passende gen-sammensætning. ♦